ISSR, Inter simple sequence repeat
Princip
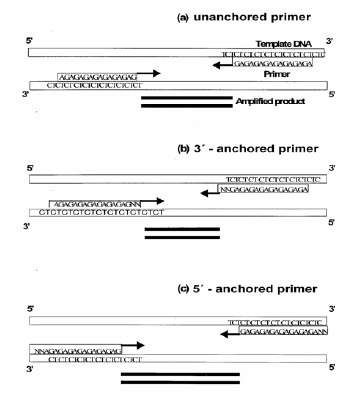
Jedná se o metodu založenou na PCR, která využívá mikrosatelitové sekvence jako primery (16-25 bp). Mikrosatelity (SSR) jsou jednoduché krátké opakující se sekvence několika nukleotidů (1-4 báze), tzv. repetice. Tyto repetitivní jednotky se vyskytují často, náhodně, v celém genomu u všech eukaryot a liší se počtem opakování. Během PCR dochází k amplifikaci úseků DNA mezi dvěmi stejnými mikrosatelitovými repetitivními sekvencemi, které jsou umístěny v řetězci DNA v opačném směru. Výsledkem jsou různě dlouhé úseky DNA mezi mikrosatelity.
Technické provedení této metody je podobné jako u RAPD analýzy (RAPD – random amplified polymorphic DNA; metody sledující délku fragmentů DNA amplifikovaných pomocí určitého krátkého náhodně zvoleného primeru), kdy jsou využity tzv. arbitrární primery. Jde tedy o metodu univerzální, nevyžadující žádné znalosti o genomu zkoumaného organismu. ISSR nachází využití při studiu genetické diverzity, fylogeneze, mapování genomu a v evoluční biologii.
ISSR primery nesou sekvenční motiv komplementární k sekvenci mikrosatelitů. Na jejich konci bývá často připojena „kotva“, tzv. anchor motiv. Nejčastěji 1-3 báze odlišné od repetitivní mikrosatelitní sekvence, které při PCR amplifikaci zajistí specifické nasednutí na konec komplementárního templátového mikrosatelitního motivu.
Vlastní postup zahrnuje běžnou PCR za využití obvykle jednoho primeru. Méně často se používá kombinace dvou ISSR primerů. Produkty PCR amplifikace jsou vizualizovány na běžném agarózovém gelu. Výsledná sada vzniklých fragmentů odráží variabilitu v distribuci a délce mikrosatelitů v genomu. Analýzu každého vzorku je nutné provést ve dvou nezávislých PCR a do statistického hodnocení zahrnout pouze „proužky“, které se objeví v obou těchto nezávislých opakováních.
Postup
PCR amplifikace- Připravíme PCR směs pro příslušný počet vzorků + 1 rezerva jako negativní kontrola. Pracujeme stále na ledu. Pro 1 reakci o objemu 20 µl je složení následující:
- PCR voda .................................... 3,8 µl
- primer (koncentrace 2,5 pM)....... 4,8 µl
- PCR enhancer ............................. 0,4 µl
- Plain PP Master Mix..................... 10 µl
- Promícháme, krátce centrifugujeme a pipetujeme po 19 µl do PCR stripů. Přidáme po 1 µl DNA vzorku. Promícháme poklepnutím prstem na strip a krátce centrifugujeme na stolní centrifuze.
- Vložíme do termocykleru a spustíme odpovídající teplotní program s použitím tzv. touchdown protokolu.
- Pipravíme 1,8 % agarózový gel.
- P skončení programu stripy vyndáme z cykleru a krátce centrifugujeme.
- Smícháme 1 µl LB s 3 µl PCR produktu, promícháme a naneseme na gel.
- Jako žebříček použijeme 4 µl 100 bp Ladderu (před a za vzorky).
- Napětí nastavíme na 40 V, po vyjetí vzorků z jamek zvýšíme na 80 V, elektroforézu necháme běžet cca 4 h.
- Pořizujeme dvě fotky gelu, první na obvyklou expozici a druhou přeexponovanou (vyniknou i slabší, tj. méně amplifikované produkty).